DNA N6-甲基腺嘌呤(N6-methyladenine, 6mA)已被报道参与真核生物的转录调控等关键生物学过程[1-6]。然而,6mA与转录的关系在不同真核生物中表现出明显差异,其参与转录调控的具体机制尚未充分阐明。嗜热四膜虫(Tetrahymena thermophila)作为一种重要的单细胞真核模式生物,已被报道含有较高的6mA水平,由甲基化酶AMT1(Adenine Methyltran sferase 1)催化,且6mA与转录存在正相关关系,是研究6mA与转录调控机制的理想体系。
2025年1月,原生动物学研究室高珊课题组在Nucleic Acids Research杂志发表题为“Methyl-dependent auto-regulation of the DNA N6-adenine methyltransferase AMT1 in the unicellular eukaryote Tetrahymena thermophila”的研究成果。这项研究以四膜虫为模式材料,发现了6mA甲基化酶AMT1的自调控和转录调控机制,并且探究了其在接合生殖时期的动态分布及生理功能,为6mA作为表观遗传标记在真核生物中参与转录调控和其他生理功能调控提供了重要线索,为未来进一步探索真核生物中6mA的功能调控机制奠定了理论基础。

团队前期以四膜虫为研究材料,鉴定出AMT1是四膜虫的主要6mA甲基化酶[7]。AMT1基因缺失会造成细胞生长缓慢,且伸缩泡异常增大。然而,AMT1如何参与基因表达调控,从而影响细胞生长发育的分子机制仍未知。
该工作发现AMT1关键酶活位点突变后不仅导致全基因组6mA水平的显著下降(图1A-C),自身基因的mRNA水平和蛋白水平也显著下降(图1D-E)。通过对AMT1基因的染色质环境进行分析发现,其具有典型的6mA分布偏好性特征,包括:稳定的核小体占位,高水平的H3K4me3和H2A.Z(图1F),以及相对较高的6mA水平(图1G)。基于此,推测AMT1基因自身的转录水平可能受6mA调控。
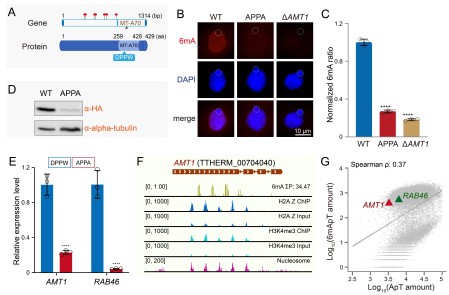
图1.AMT1关键酶活位点突变后AMT1自身的mRNA水平,蛋白水平以及表观遗传标记的分布情况。(A)AMT1的基因和蛋白模型。(B)AMT1突变后的6mA 免疫荧光染色结果。(C)AMT1突变后的6mA质谱结果。(D)AMT1突变后AMT1(HA)的蛋白印迹结果。(E)AMT1突变后AMT1基因的mRNA表达水平。(F)AMT1基因的6mA、H2A.Z、H3K4me3及核小体分布。(G)所有基因的6mA水平。
进一步地,该工作对AMT1突变株系进行了单碱基分辨率的SMRT-CCS测序。结果发现,AMT1突变后6mA水平显著下降(图2A-B),与上述6mA免疫荧光染色和质谱结果一致。更为重要的是,AMT1基因上的6mA水平明显降低(图2D-F)。转录组测序也显示,AMT1基因的mRNA表达水平随之下降(图2D)。上述结果表明AMT1可能是通过其催化活性调控自身基因的6mA水平,进一步影响其转录水平。同时,该工作利用异位过表达野生型AMT1蛋白的拯救实验,发现野生型AMT1的回补可以回升AMT1基因的6mA水平和mRNA水平(图2G)。上述结果证实了AMT1催化的6mA可调控自身的转录水平,形成了一个正反馈的转录激活机制。
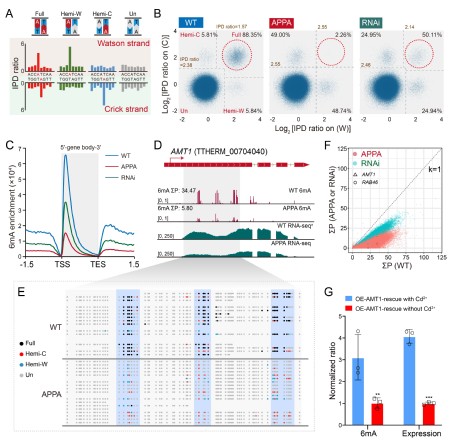
图2.AMT1突变和敲降后的6mA水平分析。(A)6mA甲基化模式。(B)AMT1突变和敲降后的6mA甲基化模式。(C)AMT1突变和敲降后的整体6mA分布情况。(D)AMT1基因的6mA分布和mRNA表达情况。(E)AMT1突变后AMT1基因的6mA 单分子模式。(F)AMT1突变和敲降后所有基因的6mA水平变化。(G)拯救细胞中AMT1基因的6mA和mRNA表达水平。
基于6mA与转录存在正相关关系,该工作还整合分析了AMT1敲除、突变、敲降三种细胞的SMRT-CCS和转录组数据,鉴定出141个高置信的AMT1-6mA依赖的基因。这些基因主要参与调控氧化还原酶活性,代谢以及跨膜转运等通路,解释了AMT1损伤后导致的生长减缓以及伸缩泡异常增大等表型。这些发现为6mA参与转录调控提供了更多证据,表明6mA可能作为一种转录激活的表观遗传标记,参与细胞内生理过程的调控(图3)。
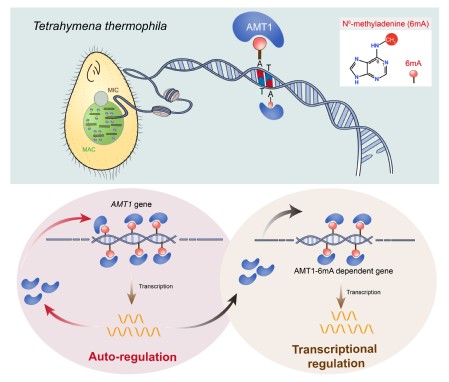
图3. AMT1自调控和转录调控的模式图。
该研究由中国海洋大学原生动物学研究室高珊课题组完成。高珊课题组博士生毕业生段丽丽和博士生李海程为该文章的共同第一作者。王媛媛副教授和高珊教授为文章的通讯作者。日本基础研究所的Kensuke Kataoka助理教授,中国海洋大学医药学院宋妮老师和原生动物学研究室马洪钢老师,高珊课题组硕士生鞠艾利、博士生张喆、牛俊骅、张雨苗、刁静涵和博士后刘永强对本文亦有重要贡献。
原文链接:
https://doi.org/10.1093/nar/gkaf022
参考文献
1. Cummings, D.J., Tait, A. and Goddard, J.M. (1974) Methylated bases in DNA from Paramecium aurelia. Biochim. Biophys. Acta., 374, 1-11.
2. Fu, Y., Luo, G.Z., Chen, K., Deng, X., Yu, M., Han, D., Hao, Z., Liu, J., Lu, X., Dore, L.C. et al. (2015) N6-methyldeoxyadenosine marks active transcription start sites in Chlamydomonas. Cell, 161, 879-892.
3. Gorovsky, M.A., Hattman, S. and Pleger, G.L. (1973) N6-methyl adenine in the nuclear DNA of a eucaryote, Tetrahymena pyriformis. J. Cell Biol., 56, 697-701.
4. Greer, E.L., Blanco, M.A., Gu, L., Sendinc, E., Liu, J., Corrales, D.A., Hsu, C.H., Aravind, L., He, C. and Shi, Y. (2015) DNA methylation on N6-adenine in C. elegans. Cell, 161, 868-878.
5. Liang, Z., Shen, L., Cui, X., Bao, S. and Yu, H. (2018) DNA N6-adenine methylation in Arabidopsis thaliana. Dev. Cell, 45, 406-416.e403.
6. Pan, B., Ye, F., Li, T., Wei, F., Warren, A., Wang, Y. and Gao, S. (2023) Potential role of N6-adenine DNA methylation in alternative splicing and endosymbiosis in Paramecium bursaria. iScience, 26, 106676.
7. Wang, Y., Sheng, Y., Liu, Y., Zhang, W., Cheng, T., Duan, L., Pan, B., Qiao, Y., Liu, Y. and Gao, S. (2019) A distinct class of eukaryotic MT-A70 methyltransferases maintain symmetric DNA N6-adenine methylation at the ApT dinucleotides as an epigenetic mark associated with transcription. Nucleic Acids Res., 47, 11771-11789.


